SOE pcr
Moderator: ArcherBarry
- Berichten: 252
SOE pcr
Hallo allemaal,
Ik snap iets niet goed aan de SOE (splicing by overlap extension) pcr.
Na een eerste PCR reactie, waarbij aan de 5' van de primers een staart wordt gekozen, krijgt men onder andere twee fragmenten, die aan de 5' kant een complementaire staart vertonen.
Deze twee fragmenten kunnen dus basenparen, maar dan moet een volledige ds DNA gemaakt worden met behulp van DNA polymerase. Aangezien deze in de 5'-->3' richting werkt, gaat dit volgens mij nog niet in deze stap, maar moet eerst nog een PCR optreden waarbij de twee speciale fragmenten met hun staart gekopieerd worden, zodat er nu dus ook twee fragmenten zijn met aan hun 3' kant een complementaire staart. Deze kunnen basenparen, DNA polymerase kan dan nucleotiden inbouwen en deze dubbelstreng kan dan geamplificeerd worden met een laatste PCR.
Nochtans vind ik overal dat je maar 1 PCR nodig hebt, dus dat het gedeelte dat ik in het rood heb aangeduid niet moet gebeuren. Kan iemand mij dit uitleggen?
Ik snap iets niet goed aan de SOE (splicing by overlap extension) pcr.
Na een eerste PCR reactie, waarbij aan de 5' van de primers een staart wordt gekozen, krijgt men onder andere twee fragmenten, die aan de 5' kant een complementaire staart vertonen.
Deze twee fragmenten kunnen dus basenparen, maar dan moet een volledige ds DNA gemaakt worden met behulp van DNA polymerase. Aangezien deze in de 5'-->3' richting werkt, gaat dit volgens mij nog niet in deze stap, maar moet eerst nog een PCR optreden waarbij de twee speciale fragmenten met hun staart gekopieerd worden, zodat er nu dus ook twee fragmenten zijn met aan hun 3' kant een complementaire staart. Deze kunnen basenparen, DNA polymerase kan dan nucleotiden inbouwen en deze dubbelstreng kan dan geamplificeerd worden met een laatste PCR.
Nochtans vind ik overal dat je maar 1 PCR nodig hebt, dus dat het gedeelte dat ik in het rood heb aangeduid niet moet gebeuren. Kan iemand mij dit uitleggen?
- Berichten: 3.963
Re: SOE pcr
Ik snap niet precies waarom dat niet zou lukken.
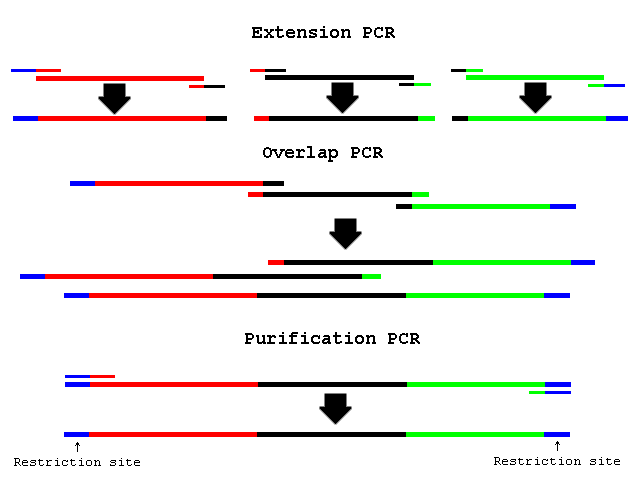
(Bron: http://openwetware.org/wiki/PCR_Overlap_Extension)
Bij de Overlap-PCR kan je toch je primer designen tegen de restrictiesite (blauw) en dan gaat het polymerase toch over alles heen?
Finaal doe je wel best nog een extra (3de) PCR om alle fragmenten van restrictiesite tot restrictiesite te amplificeren zodat je het juiste fragment op gel kan isoleren.

(Bron: http://openwetware.org/wiki/PCR_Overlap_Extension)
Bij de Overlap-PCR kan je toch je primer designen tegen de restrictiesite (blauw) en dan gaat het polymerase toch over alles heen?
Finaal doe je wel best nog een extra (3de) PCR om alle fragmenten van restrictiesite tot restrictiesite te amplificeren zodat je het juiste fragment op gel kan isoleren.
"Success is the ability to go from one failure to another with no loss of enthusiasm" - Winston Churchill
- Berichten: 252
Re: SOE pcr
Ik ging er vanuit dat de eerste PCR maar uit 1 cyclus bestond, maar uiteraard gaat het hier om een 30tal cycli. In dat geval is er dus ook een complementaire staart aan de 3' kant en is mijn probleem opgelost!Kravitz schreef: ↑za 21 dec 2013, 10:07
Ik snap niet precies waarom dat niet zou lukken.
(Bron: http://openwetware.o...erlap_Extension)
Bij de Overlap-PCR kan je toch je primer designen tegen de restrictiesite (blauw) en dan gaat het polymerase toch over alles heen?
Finaal doe je wel best nog een extra (3de) PCR om alle fragmenten van restrictiesite tot restrictiesite te amplificeren zodat je het juiste fragment op gel kan isoleren.
Bedankt